一、Tăng độ nhạy của hệ thống phản ứng:
1. Phân lập RNA chất lượng cao:
Quá trình tổng hợp cDNA thành công đến từ RNA chất lượng cao.RNA chất lượng cao ít nhất phải có chiều dài đầy đủ và không có chất ức chế men phiên mã ngược như EDTA hoặc SDS.Chất lượng của RNA xác định lượng thông tin trình tự tối đa mà bạn có thể phiên mã thành cDNA.Phương pháp tinh sạch RNA phổ biến là phương pháp một bước sử dụng guanidine isothiocyanate/axit phenol.Để ngăn ngừa ô nhiễm bởi một lượng nhỏ RNase, RNA được phân lập từ các mẫu giàu RNase (chẳng hạn như tuyến tụy) cần được bảo quản trong formaldehyde để bảo quản RNA chất lượng cao, đặc biệt là để bảo quản lâu dài.RNA chiết xuất từ gan chuột về cơ bản đã bị phân hủy sau khi được bảo quản trong nước trong một tuần, trong khi RNA chiết xuất từ lá lách chuột vẫn ổn định sau khi được bảo quản trong nước trong 3 năm.Ngoài ra, các bản ghi dài hơn 4 kb nhạy cảm hơn với sự xuống cấp của RNase theo dõi so với các bản ghi nhỏ.Để tăng tính ổn định của các mẫu RNA được lưu trữ, RNA có thể được hòa tan trong formamide đã khử ion và được bảo quản ở -70°C.Formamide được sử dụng để bảo quản RNA phải không có mảnh vụn phân hủy RNA.RNA từ tuyến tụy có thể được bảo quản trong formamide ít nhất một năm.Khi chuẩn bị sử dụng RNA, bạn có thể sử dụng phương pháp sau để kết tủa RNA: thêm NaCl với 0,2M và thể tích ethanol gấp 4 lần, đặt ở nhiệt độ phòng trong 3-5 phút và ly tâm ở 10.000 × g trong 5 phút.
2. Sử dụng men phiên mã ngược RNaseH-không hoạt động (RNaseH-):
Các chất ức chế RNase thường được thêm vào các phản ứng phiên mã ngược để tăng thời gian và năng suất tổng hợp cDNA.Chất ức chế RNase nên được thêm vào trong phản ứng tổng hợp chuỗi đầu tiên với sự có mặt của chất đệm và chất khử (chẳng hạn như DTT), bởi vì quá trình trước khi tổng hợp cDNA làm biến tính chất ức chế, do đó giải phóng RNase liên kết có thể làm suy giảm RNA.Các chất ức chế protein RNase chỉ ngăn chặn sự phân hủy RNA bởi RNase A, B, C và không ngăn chặn RNase trên da, vì vậy hãy cẩn thận không đưa RNase từ ngón tay của bạn mặc dù đã sử dụng các chất ức chế này.
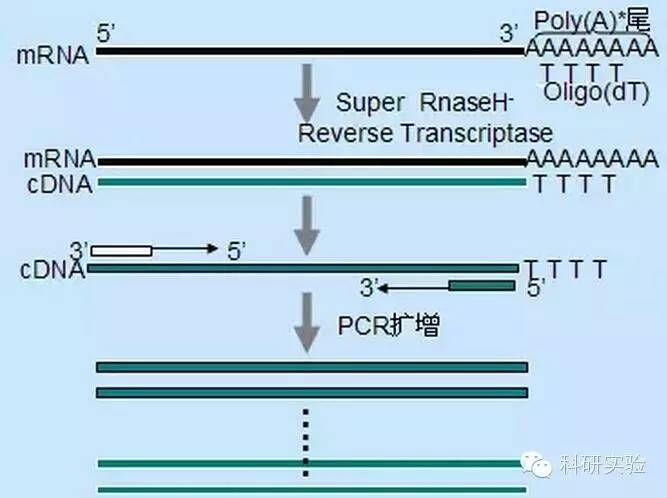
Enzyme phiên mã ngược xúc tác quá trình chuyển đổi RNA thành cDNA.Cả M-MLV và AMV đều có hoạt tính RNaseH nội sinh ngoài hoạt tính polymerase của chính chúng.Hoạt tính RNaseH và hoạt tính polymerase cạnh tranh với nhau để giành được chuỗi lai tạo giữa khuôn mẫu RNA và đoạn mồi DNA hoặc chuỗi mở rộng cDNA, đồng thời làm suy giảm chuỗi RNA trong phức hợp RNA:DNA.Khuôn mẫu RNA bị suy giảm do hoạt động của RNaseH không còn có thể đóng vai trò là chất nền hiệu quả để tổng hợp cDNA, điều này làm giảm năng suất và thời gian tổng hợp cDNA.Do đó, sẽ có lợi nếu loại bỏ hoặc giảm đáng kể hoạt động RNaseH của enzyme phiên mã ngược.。
SuperScript Ⅱ phiên mã ngược, RNaseH- MMLV phiên mã ngược và phiên mã ngược thermoScript, RNaseH- AMV, có thể thu được nhiều cDNA hơn và có độ dài đầy đủ hơn so với MMLV và AMV.Độ nhạy của RT-PCR sẽ bị ảnh hưởng bởi số lượng tổng hợp cDNA.ThermoScript nhạy hơn nhiều so với AMV.Kích thước của các sản phẩm RT-PCR bị giới hạn bởi khả năng tổng hợp cDNA của enzyme phiên mã ngược, đặc biệt là khi nhân bản các cDNA lớn hơn.So với MMLV, SuperScripⅡ tăng đáng kể năng suất của các sản phẩm RT-PCR dài.Enzyme phiên mã ngược RNaseH cũng tăng khả năng điều nhiệt, do đó phản ứng có thể được thực hiện ở nhiệt độ cao hơn nhiệt độ bình thường 37-42°C.Trong các điều kiện tổng hợp được đề xuất, hãy sử dụng mồi oligo(dT) và 10 μCi của [α-P]dCTP.Tổng sản lượng của sợi đầu tiên được tính toán bằng phương pháp kết tủa TCA.CDNA có chiều dài đầy đủ được phân tích bằng cách sử dụng các dải được sắp xếp theo kích thước được cắt bỏ và tính trên gel agarose kiềm.
3. Tăng nhiệt độ ủ để phiên mã ngược:
Nhiệt độ ủ cao hơn giúp mở cấu trúc thứ cấp RNA, làm tăng năng suất của phản ứng.Đối với hầu hết các mẫu RNA, việc ủ RNA và đoạn mồi ở 65°C mà không có dung dịch đệm hoặc muối, sau đó làm lạnh nhanh trên đá sẽ loại bỏ hầu hết các cấu trúc thứ cấp và cho phép các đoạn mồi liên kết với nhau.Tuy nhiên, một số mẫu vẫn có cấu trúc thứ cấp, ngay cả sau khi biến tính nhiệt.Việc khuếch đại các mẫu khó này có thể được thực hiện bằng cách sử dụng ThermoScript Reverse Transcriptase và đặt phản ứng sao chép ngược ở nhiệt độ cao hơn để cải thiện khả năng khuếch đại.Nhiệt độ ủ cao hơn cũng có thể làm tăng tính đặc hiệu, đặc biệt là khi các đoạn mồi đặc hiệu cho gen (GSP) được sử dụng để tổng hợp cDNA (xem Chương 3).Nếu sử dụng GSP, hãy đảm bảo rằng Tm của mồi giống với nhiệt độ ủ dự kiến.Không sử dụng mồi oligo(dT) và ngẫu nhiên trên 60°C.Mồi ngẫu nhiên yêu cầu ủ ở 25°C trong 10 phút trước khi tăng lên 60°C.Ngoài việc sử dụng nhiệt độ phiên mã ngược cao hơn, tính đặc hiệu cũng có thể được cải thiện bằng cách chuyển trực tiếp hỗn hợp RNA/mồi từ nhiệt độ biến tính 65°C sang nhiệt độ ủ phiên mã ngược và thêm hỗn hợp phản ứng 2× đã được làm ấm trước (tổng hợp bắt đầu nóng cDNA) .Cách tiếp cận này giúp ngăn chặn sự ghép đôi cơ sở liên phân tử xảy ra ở nhiệt độ thấp hơn.Có thể đơn giản hóa việc chuyển đổi nhiều nhiệt độ cần thiết cho RT-PCR bằng cách sử dụng máy luân nhiệt.
Tth polymerase bền nhiệt hoạt động như một DNA polymerase với sự có mặt của Mg2+ và như một RNA polymerase với sự có mặt của Mn2+.Nó có thể được giữ ấm ở nhiệt độ tối đa là 65°C.Tuy nhiên, sự hiện diện của Mn2+ trong quá trình PCR làm giảm độ trung thực, điều này làm cho Tth polymerase ít phù hợp hơn cho quá trình khuếch đại có độ chính xác cao, chẳng hạn như tạo dòng cDNA.Ngoài ra, Tth có hiệu suất phiên mã ngược thấp, làm giảm độ nhạy, và do phiên mã ngược và PCR có thể được thực hiện với một loại enzyme duy nhất, nên không thể sử dụng các phản ứng kiểm soát mà không có phiên mã ngược để so sánh các sản phẩm khuếch đại cDNA với DNA bộ gen gây ô nhiễm.Các sản phẩm khuếch đại đã được tách ra.
4. Phụ gia thúc đẩy phiên mã ngược:
Các chất phụ gia bao gồm glycerol và DMSO được thêm vào phản ứng tổng hợp chuỗi thứ nhất, có thể làm giảm tính ổn định của chuỗi kép axit nucleic và tháo gỡ cấu trúc thứ cấp của RNA.Có thể thêm tối đa 20% glycerol hoặc 10% DMSO mà không ảnh hưởng đến hoạt động của SuperScript II hoặc MMLV.AMV cũng có thể dung nạp tới 20% glycerol mà không bị mất hoạt tính.Để tối đa hóa độ nhạy của RT-PCR trong phản ứng sao chép ngược SuperScriptⅡ, 10% glycerol có thể được thêm vào và ủ ở 45°C.Nếu cho 1/10 sản phẩm của phản ứng sao chép ngược vào PCR thì nồng độ glycerol trong phản ứng khuếch đại là 0,4% không đủ ức chế PCR.
5. Xử lý RNaseH:
Xử lý các phản ứng tổng hợp cDNA bằng RNaseH trước PCR có thể làm tăng độ nhạy.Đối với một số mẫu, người ta cho rằng RNA trong phản ứng tổng hợp cDNA ngăn chặn sự gắn kết của các sản phẩm khuếch đại, trong trường hợp đó, xử lý RNaseH có thể làm tăng độ nhạy.Nói chung, việc xử lý RNaseH là cần thiết khi khuếch đại các mẫu mục tiêu cDNA có độ dài đầy đủ dài hơn, chẳng hạn như bệnh xơ cứng củ II bản sao thấp.Đối với mẫu khó này, xử lý RNaseH đã tăng cường tín hiệu do cDNA tổng hợp bởi SuperScript II hoặc AMV tạo ra.Đối với hầu hết các phản ứng RT-PCR, việc xử lý RNaseH là tùy chọn, vì bước biến tính PCR ở 95°C thường thủy phân RNA trong phức hợp RNA:DNA.
6. Cải thiện phương pháp phát hiện RNA nhỏ:
RT-PCR đặc biệt khó khăn khi chỉ có một lượng nhỏ RNA.Glycogen được thêm vào làm chất mang trong quá trình phân lập RNA giúp tăng năng suất của các mẫu nhỏ.RNase-free glycogen có thể được bổ sung cùng lúc với việc bổ sung Trizol.Glycogen hòa tan trong nước và có thể được giữ trong pha nước với RNA để hỗ trợ quá trình kết tủa tiếp theo.Đối với các mẫu có ít hơn 50 mg mô hoặc 106 tế bào nuôi cấy, nồng độ glycogen không chứa RNase được khuyến nghị là 250 μg/ml.
Việc thêm BSA acetyl hóa vào phản ứng sao chép ngược bằng SuperScript II có thể làm tăng độ nhạy và đối với một lượng nhỏ RNA, việc giảm lượng SuperScript II và thêm 40 đơn vị chất ức chế nuclease RNaseOut có thể làm tăng mức độ phát hiện.Nếu sử dụng glycogen trong quá trình phân lập RNA, vẫn nên bổ sung chất ức chế BSA hoặc RNase khi sử dụng SuperScript II cho phản ứng phiên mã ngược.
二、Tăng độ đặc hiệu của RT-PCR
1. Tổng hợp CND:
Quá trình tổng hợp cDNA sợi đầu tiên có thể được bắt đầu bằng ba phương pháp khác nhau, tính đặc hiệu tương đối của chúng ảnh hưởng đến số lượng và loại cDNA được tổng hợp.
Phương pháp mồi ngẫu nhiên là phương pháp ít cụ thể nhất trong ba phương pháp.Các đoạn mồi ủ tại nhiều vị trí trong suốt bản phiên mã, tạo ra các cDNA ngắn, có độ dài một phần.Phương pháp này thường được sử dụng để thu được 5′ trình tự kết thúc và thu được cDNA từ các mẫu RNA có vùng cấu trúc bậc hai hoặc có vị trí kết thúc không thể sao chép bằng enzym phiên mã ngược.Để thu được cDNA dài nhất, tỷ lệ đoạn mồi so với RNA trong mỗi mẫu RNA cần được xác định theo kinh nghiệm.Nồng độ ban đầu của mồi ngẫu nhiên nằm trong khoảng từ 50 đến 250 ng trên 20 μl phản ứng.Vì cDNA được tổng hợp từ RNA tổng số bằng cách sử dụng đoạn mồi ngẫu nhiên chủ yếu là RNA ribosome, poly(A)+RNA thường được chọn làm khuôn mẫu.
Các đoạn mồi Oligo(dT) cụ thể hơn so với các đoạn mồi ngẫu nhiên.Nó lai với đuôi poly(A) được tìm thấy ở đầu 3′ của hầu hết các mARN của sinh vật nhân chuẩn.Bởi vì poly(A)+ RNA chiếm khoảng 1% đến 2% tổng số RNA, nên số lượng và độ phức tạp của cDNA ít hơn nhiều so với các đoạn mồi ngẫu nhiên.Do tính đặc hiệu cao nên nhìn chung oligo(dT) không yêu cầu tối ưu hóa tỷ lệ RNA so với mồi và chọn lọc poly(A)+.Nên sử dụng 0,5μg oligo(dT) cho mỗi hệ thống phản ứng 20μl.oligo(dT)12-18 phù hợp với hầu hết RT-PCR.Hệ thống RT-PCR ThermoScript cung cấp oligo(dT)20 vì tính ổn định nhiệt tốt hơn cho nhiệt độ ủ cao hơn.
Đoạn mồi đặc hiệu gen (GSP) là đoạn mồi đặc hiệu nhất cho bước phiên mã ngược.GSP là một oligonucleotide đối nghĩa có thể lai cụ thể với trình tự đích RNA, không giống như các đoạn mồi hoặc oligo(dT) ngẫu nhiên, gắn với tất cả các RNA.Các quy tắc tương tự được sử dụng để thiết kế đoạn mồi PCR áp dụng cho việc thiết kế GSP trong các phản ứng phiên mã ngược.GSP có thể có cùng trình tự với đoạn mồi khuếch đại gắn vào đầu 3′ của mARN hoặc GSP có thể được thiết kế để gắn vào xuôi dòng của đoạn mồi khuếch đại ngược.Đối với một số đối tượng được khuếch đại, cần phải thiết kế nhiều hơn một đoạn mồi antisense để RT-PCR thành công vì cấu trúc thứ cấp của RNA đích có thể ngăn cản sự gắn kết của đoạn mồi.Nên sử dụng 1 pmol antisense GSP trong phản ứng tổng hợp chuỗi đầu tiên 20 μl.
2. Tăng nhiệt độ ủ để phiên mã ngược:
Để tận dụng toàn bộ lợi thế của tính đặc hiệu của GSP, nên sử dụng enzyme phiên mã ngược có khả năng điều nhiệt cao hơn.Enzyme phiên mã ngược ổn định nhiệt có thể được ủ ở nhiệt độ cao hơn để tăng tính nghiêm ngặt của phản ứng.Ví dụ: nếu ủ GSP ở 55°C, tính đặc hiệu của GSP sẽ không được sử dụng đầy đủ nếu AMV hoặc M-MLV được sử dụng để sao chép ngược ở mức nghiêm ngặt thấp là 37°C.Tuy nhiên, SuperScript II và ThermoScript có thể được phản ứng ở 50°C hoặc cao hơn, điều này sẽ loại bỏ các sản phẩm không đặc hiệu được tạo ra ở nhiệt độ thấp hơn.Để có độ đặc hiệu tối đa, hỗn hợp RNA/mồi có thể được chuyển trực tiếp từ nhiệt độ biến tính 65°C sang nhiệt độ ủ phiên mã ngược và thêm vào hỗn hợp phản ứng 2× đã được làm ấm trước (khởi động nóng tổng hợp cDNA).Điều này giúp ngăn chặn sự ghép nối cơ sở liên phân tử ở nhiệt độ thấp.Có thể đơn giản hóa nhiều quá trình chuyển đổi nhiệt độ cần thiết cho RT-PCR bằng cách sử dụng máy luân nhiệt.
3. Giảm ô nhiễm DNA bộ gen:
Một khó khăn tiềm ẩn gặp phải với RT-PCR là sự nhiễm bẩn DNA bộ gen trong RNA.Sử dụng phương pháp phân lập RNA tốt, chẳng hạn như Thuốc thử Trizol, sẽ làm giảm lượng DNA bộ gen nhiễm vào quá trình chuẩn bị RNA.Để tránh các sản phẩm có nguồn gốc từ DNA bộ gen, RNA có thể được xử lý bằng DNase I cấp độ khuếch đại để loại bỏ DNA gây ô nhiễm trước khi phiên mã ngược.Quá trình tiêu hóa DNase I kết thúc bằng cách ủ các mẫu trong 2,0 mM EDTA trong 10 phút ở 65°C.EDTA có thể chelate các ion magie, ngăn chặn quá trình thủy phân RNA phụ thuộc ion magie ở nhiệt độ cao.
Để tách cDNA được khuếch đại khỏi các sản phẩm khuếch đại DNA bộ gen gây nhiễm bẩn, các đoạn mồi có thể được thiết kế sao cho mỗi đoạn mồi gắn với các exon riêng biệt.Các sản phẩm PCR có nguồn gốc từ cDNA sẽ ngắn hơn các sản phẩm PCR có nguồn gốc từ DNA bộ gen bị ô nhiễm.Ngoài ra, một thử nghiệm đối chứng không có phiên mã ngược đã được thực hiện trên mỗi mẫu RNA để xác định xem một đoạn đã cho có nguồn gốc từ DNA bộ gen hay cDNA hay không.Sản phẩm PCR thu được không qua phiên mã ngược có nguồn gốc từ bộ gen.
Thời gian đăng: 16-05-2023








